OMS: pandemia en América Latina “apenas comienza”
El representante de la Organización Mundial de la Salud (OMS) y la Organización Panamericana de la Salud (OPS) en El Salvador, Carlos Garzón, advirtió que la pandemia de COVID-19 “apenas comienza” en la región, y sugirió evaluar esto antes de pensar en una apertura económica.
“En la medida en que se empiezan a escuchar noticias sobre la apertura de Europa, tomamos en cuenta cómo está creciendo esta enfermedad en las Américas. Estamos cinco semanas atrás de Europa, allá están pasando los picos altos de la enfermedad y aquí a penas estamos comenzando”, aseguró Garzón, en conferencia de prensa con el Gabinete de Salud.
Según el especialista, antes de pensar en la apertura, “es necesario tener en cuenta que los servicios de salud sean suficientes y estén disponibles para poder atender a las personas” así como “educación comunitaria suficiente” para mantener medidas de protección.
Garzón advirtió que los países en América Latina “están llegando al límite de la capacidad de atención con el crecimiento tan rápido del número de atenciones”, a consecuencia de no conservar las medidas de aislamiento social y cuarentena, por lo que “el número de contagios comunitarios empiezan a aumentar de manera rápida”.
Panamá es el país centroamericano con más contagios de Covid-19
El Ministerio de Salud de Panamá informó que en el país se registran 7.090 casos confirmados del nuevo coronavirus y un total de 197 fallecidos, lo que la convierte en la nación con más casos de Covid-19 en Centroamérica.
Durante la conferencia de prensa del sábado, autoridades sanitarias panameñas advirtieron que es preocupante el poco descenso de las cifras, así como el incumplimiento de la cuarentena decretada.
En este sentido, el viceministro de Salud, Luis Francisco Sucre, aseguró que “no estamos logrando los objetivos requeridos para esta etapa», por lo que alertó que, de mantenerse la situación, se tendrá que postergar la apertura de algunos sectores panameños.
Compartimos la actualización de datos sobre el #COVID19 en nuestro país.
Sábado 2 de mayo de 2020.#ProtégetePanamá#UnidosVenceremos pic.twitter.com/eEGuvZv6tT— Ministerio de Salud de Panamá (@MINSAPma) May 2, 2020
Por su parte, el presidente de Panamá, Laurentino Cortizo, se pronunció sobre un posible levantamiento paulatino de la cuarentena, así como la reactivación del país para instaurar una “nueva normalidad” a partir de mayo, pese a que estiman un pico de las cifras de contagiados para los próximos días.
En tanto, expertos advierten que el impacto social y económico del virus se ha visto agravado por el manejo de la pandemia en el país, donde más de 90.000 trabajadores han sido afectados por la suspensión de contratos desde marzo, fecha de inicio de la pandemia en esa nación.
Asimismo, señalan que al instaurarse las medidas de distanciamiento social, cerca del 50 por ciento restante de los trabajadores panameños han evidenciado daños a sus economías personales y familiares.
Un equipo de científicos de los departamentos de Virología y Biotecnología, y Genómica y Proteómica del Instituto Conmemorativo Gorgas de Estudios de la Salud (Icges) comenzó a obtener respuestas sobre el linaje u origen del virus SARS-CoV-2, así como de su llegada al país.
La secuenciación genética hecha a muestras de pacientes obtenidas durante tres semanas (del 9 al 30 de marzo) ha permitido establecer que el paciente cero estaba en Panamá en el mes de febrero, pero todavía no han logrado identificarlo.
Alexander Martínez, jefe del departamento de Genómica y Proteómica, reveló que han secuenciado el virus de 120 muestras de pacientes, lo que les ha permitido obtener 110 genomas completos. En el resto de las muestras no se logró secuenciar el genoma completo, solo parcial. Pero el trabajo no termina allí. Siguen realizando análisis con un sistema de alta generación.
Los datos obtenidos en los estudios han permitido establecer que hay 8 linajes (cepas) que tienen un origen de importación y un linaje de origen local.
En ese sentido, Martínez explicó que la cepa A2 (España) que llegó al país, mutó y fijó una transmisión entre la población, originando posiblemente la cepa local A2PAN.
Añadió que los resultados preliminares, con probabilidades estadísticas de 95%, permiten establecer que el linaje local A2 PAN pudo haber llegado entre el 9 de febrero y el 4 de marzo.
Martínez afirmó que, de acuerdo con los resultados, los virus importados llegaron todos simultáneamente después de Carnaval, con los viajeros que visitaron países donde había infección activa del nuevo coronavirus.
Los investigadores siguen secuenciando el virus para conocer más sobre la transmisión entre la población panameña.
Científicos del Gorgas, decodificando el virus
Desde que se reportó el primer caso en el país, el pasado 9 de marzo, científicos del Instituto Conmemorativo Gorgas de Estudios de la Salud (Icges) no solo están realizando –a diario– las pruebas de laboratorio para confirmar nuevos casos de la enfermedad Covid-19, sino que continúan descifrando el genoma del virus SARS-CoV-2.
La secuencia del genoma es importante por cuatro razones: proporciona información independiente de los orígenes de la epidemia y nuevos linajes (cepas); permite conocer la fuente de transmisión de un infectado; brinda datos de la dinámica epidémica de enfermedades pasadas; y da luz sobre la adaptación del patógeno en la población.
Así lo planteó en el Café Científico de la Secretaría Nacional de Ciencia, Tecnología e Innovación, Alexander Martínez, jefe del departamento de Investigación en Genómica y Proteómica, quien explicó que la secuencia del genoma del virus brinda información sobre la fuente de infección, ya que se analiza el virus específico que afectó al paciente.
Además permite, luego de localizar el paciente 1 y 2, secuenciar ambos virus y obtener resultados similares, establecer un vínculo entre los pacientes; y buscar mutaciones que hacen que el virus se pueda transmitir mejor en la población, explicó el científico.
Por esas razones, este equipo del Gorgas trabaja en la secuenciación del virus y ya tiene las primeras respuestas sobre el linaje y llegada del virus al país, entre otros detalles.
110 genomas
Martínez manifestó que secuenciaron el patógeno de 120 muestras de pacientes, obtenidas durante tres semanas (del 9 al 30 de marzo, lo que les permitió obtener 110 genomas completos, y siguen realizando más análisis con un sistema de alta generación. En el resto de las muestras analizadas no se logró secuenciar el genoma completo, solo parcial.
Precisó que clasificaron a los pacientes de la siguiente forma: si conocían cómo se habían contagiado, si tenían antecedentes de un viaje, si era trabajador en la primera línea de respuesta de la pandemia, familiar de un contacto positivo o si tenía historial de viaje a un lugar con brote activo del virus SARS-CoV-2.
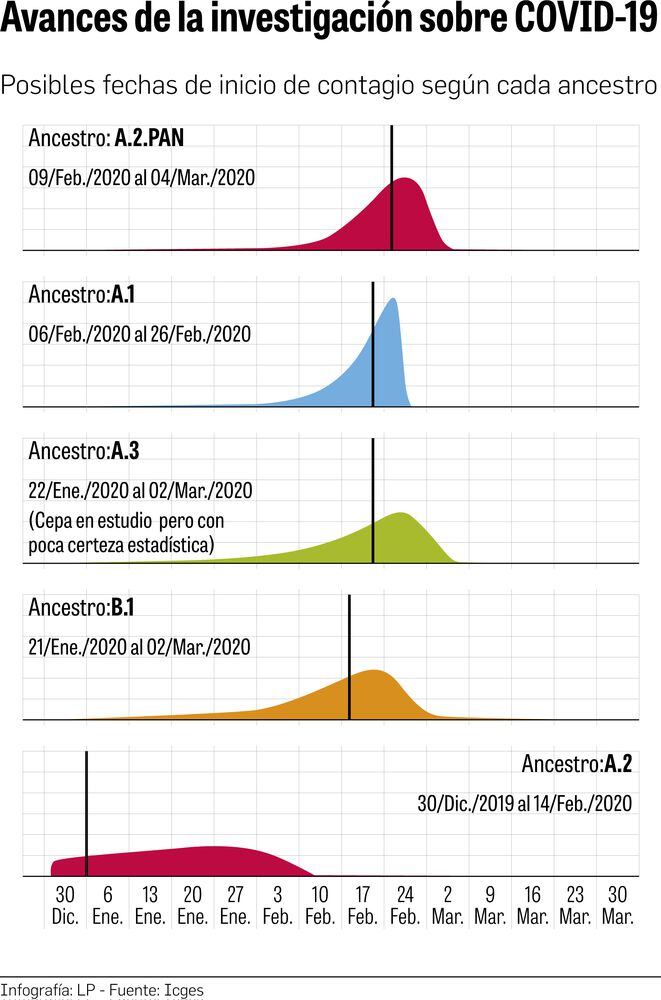
Los datos obtenidos en los estudios, subrayó Martínez, han permitido identificar 8 cepas que tienen un origen de importación (España, Italia y Estados Unidos) y una posiblemente de origen local, es decir, que llegó, se estableció y mutó fijando cadenas de transmisión efectiva en la población.
Luego de establecer el origen del linaje, el equipo de Genómica y Proteómica también buscó conocer cuándo llegó el virus a Panamá. Lo hizo mediante el análisis de ancestro más común de las cepas A1 (Estados Unidos), A2 (España), A2 PAN (origen local y probablemente descendiente de A2), B1 (Italia), y otras cepas. Es un análisis -ancestro más común- en el que se le coloca una fecha a la secuenciación y un software especializado se encarga de identificar los nodos de los ancestros.
Los resultados preliminares, con probabilidades estadísticas de 95%, permiten establecer que el linaje local A2 PAN pudo haber llegado entre el 9 de febrero y el 4 de marzo.
Otros análisis también arrojaron que la cepa A1 (Estados Unidos) llegó entre el 6 de febrero y el 26 de febrero, es decir, un poco antes del A2PAN; y la B1 (Italia), entre el 21 de enero y el 2 de marzo. (Ver gráfica).
Martínez afirmó que, de acuerdo con los resultados, el paciente cero ya estaba en Panamá en febrero, y que los virus importados llegaron todos simultáneamente después de Carnaval con los viajeros que visitaron países donde había infección activa del nuevo coronavirus.
No obstante, aclaró que siguen descifrando el virus y buscando más respuestas sobre su presencia y comportamiento en el país.
Aislamiento del virus
Sandra López Vergés, jefa del departamento de Investigación de Virología y Biotecnología del Icges, acotó que trabajan en el diagnóstico diario de nuevos casos de Covid-19 para encontrarlos, aislarlos y poder disminuir la transmisión del virus.
Además, trabajan en varias técnicas que serán muy útiles para diferentes campos de la investigación. Por el momento, dijo, los resultados obtenidos en la parte de secuenciación, filogenética y análisis del virus son muy interesantes.
La científica indicó que este virus es de la familia coronaviridae, a la que, por muchos años después de ser identificada, no se le prestó atención porque la mayoría son virus de origen veterinario, y la virología médica no se enfocaba en ellos.
Sostuvo que los pocos virus de esta familia coronaviridae que pueden afectar a humanos causan infecciones instestinales o respiratorios.
En la década de 1960 y 1970 se identificaron los primeros coronavirus respiratorios que podían infectar humanos, y los cuatro que son endémicos en el mundo entero y nuestro país causan resfriado común: 229E, OC43, NL63 y HKU1.
Los coronavirus son pequeños que infectan animales (mamíferos), sobretodo los alfacoronavirus ( 229E y NL63) y los betacoronavirus (OC43, HKU1 MERS, SARS-CoV y SARS-CoV-2), y de allí es que provienen los diferentes virus emergentes que han logrado infectar al humano, explicó López Vergés.
Señaló que todos los virus emergentes que pasaron del animal al humano han logrado adaptarse de tal manera que la transmisión de humano a humano sea eficiente. El único que no lo logró fue el MERS, el más letal.Los datos obtenidos en los estudios, subrayó Martínez, han permitido identificar 8 cepas que tienen un origen de importación (España, Italia y Estados Unidos) y una posiblemente de origen local, es decir, que llegó, se estableció y mutó fijando cadenas de transmisión efectiva en la población.
Luego de establecer el origen del linaje, el equipo de Genómica y Proteómica también buscó conocer cuándo llegó el virus a Panamá. Lo hizo mediante el análisis de ancestro más común de las cepas A1 (Estados Unidos), A2 (España), A2 PAN (origen local y probablemente descendiente de A2), B1 (Italia), y otras cepas. Es un análisis -ancestro más común- en el que se le coloca una fecha a la secuenciación y un software especializado se encarga de identificar los nodos de los ancestros.
Los resultados preliminares, con probabilidades estadísticas de 95%, permiten establecer que el linaje local A2 PAN pudo haber llegado entre el 9 de febrero y el 4 de marzo.
Otros análisis también arrojaron que la cepa A1 (Estados Unidos) llegó entre el 6 de febrero y el 26 de febrero, es decir, un poco antes del A2PAN; y la B1 (Italia), entre el 21 de enero y el 2 de marzo. (Ver gráfica).
Martínez afirmó que, de acuerdo con los resultados, el paciente cero ya estaba en Panamá en febrero, y que los virus importados llegaron todos simultáneamente después de Carnaval con los viajeros que visitaron países donde había infección activa del nuevo coronavirus.
No obstante, aclaró que siguen descifrando el virus y buscando más respuestas sobre su presencia y comportamiento en el país.
Aislamiento del virus
Sandra López Vergés, jefa del departamento de Investigación de Virología y Biotecnología del Icges, acotó que trabajan en el diagnóstico diario de nuevos casos de Covid-19 para encontrarlos, aislarlos y poder disminuir la transmisión del virus.
Además, trabajan en varias técnicas que serán muy útiles para diferentes campos de la investigación. Por el momento, dijo, los resultados obtenidos en la parte de secuenciación, filogenética y análisis del virus son muy interesantes.
La científica indicó que este virus es de la familia coronaviridae, a la que, por muchos años después de ser identificada, no se le prestó atención porque la mayoría son virus de origen veterinario, y la virología médica no se enfocaba en ellos.
Sostuvo que los pocos virus de esta familia coronaviridae que pueden afectar a humanos causan infecciones instestinales o respiratorios.
En la década de 1960 y 1970 se identificaron los primeros coronavirus respiratorios que podían infectar humanos, y los cuatro que son endémicos en el mundo entero y nuestro país causan resfriado común: 229E, OC43, NL63 y HKU1.
Los coronavirus son pequeños que infectan animales (mamíferos), sobretodo los alfacoronavirus ( 229E y NL63) y los betacoronavirus (OC43, HKU1 MERS, SARS-CoV y SARS-CoV-2), y de allí es que provienen los diferentes virus emergentes que han logrado infectar al humano, explicó López Vergés.
Señaló que todos los virus emergentes que pasaron del animal al humano han logrado adaptarse de tal manera que la transmisión de humano a humano sea eficiente. El único que no lo logró fue el MERS, el más letal.

